关于SVM其基础知识和原理已经在一篇文章中提及到了,戳这里 。
本文主要是谈谈如何利用sklearn包中的SVM来进行乳腺癌的检测。
在 sklearn中使用svm 通过以下语句即可完成一个SVM模型的创建。
1 2 3 4 5 6 7 8 9 from sklearn import svmclassfier_model = svm.SVC(C = 1.0 , kernel = 'rbf' , degree=3 , gamma='auto' ) regression_model = svm.SVR(kernel = 'rbf' , degree=3 , gamma = 'auto' )
这里重点关注SVC 的构造函数:model = svm.SVC(kernel=‘rbf’, C=1.0, gamma=‘auto’),这里有三个重要的参数 kernel、C 和 gamma。
kernel :代表核函数,默认为 rbf 高斯核函数,主要可选项有:
inear:线性核函数,在数据为线性可分时,运算速度快,效果好,无法处理线性不可分的数据。
poly:多项式核函数,可以将数据从低维空间映射到高维空间,但是参数较多,计算两大。
rbf:高斯核函数(默认),将样本映射到高维空间,但是相较于多项式核函数来说参数较少,性能不错。
sigmoid:sigmoid 核函数,当选用sigmoid,svm实现的时多层神经网络。
C 代表的是目标函数的惩罚系数,惩罚系数指的是分错样本时的惩罚程度。当 C 越大的时候,分类器的准确性越高,但同样容错率会越低,泛化能力会变差。相反,C 越小,泛化能力越强,但是准确性会降低。gamma 代表核函数的系数,默认为样本特征数的倒数,即 gamma = 1 / n_features。
然后训练和预测的方式是:
1 2 model.fit(train_X,train_y) model.predict(test_X)
使用SVM进行乳腺癌检测 首先必须确定的是,乳腺癌检测是一个分类问题。
检测存在两个过程:
数据准备阶段:
数据加载:加载数据集;
数据清洗:删除无关的列,对列数据属性进行变换或者映射;
特征选择:一般是剔除无关特征,运用降维方式,用少量特征代表数据的特性,增强分类器的泛化能力,避免数据过拟合。
数据规范化:Z-score等等
分类阶段:
创建模型
训练模型
预测模型
评估模型
整个过程的代码如下:
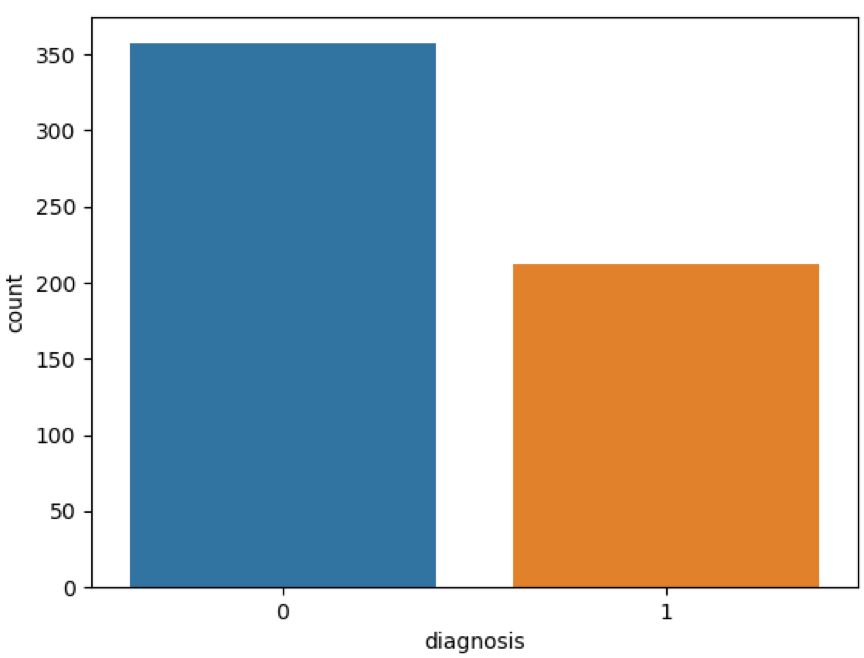
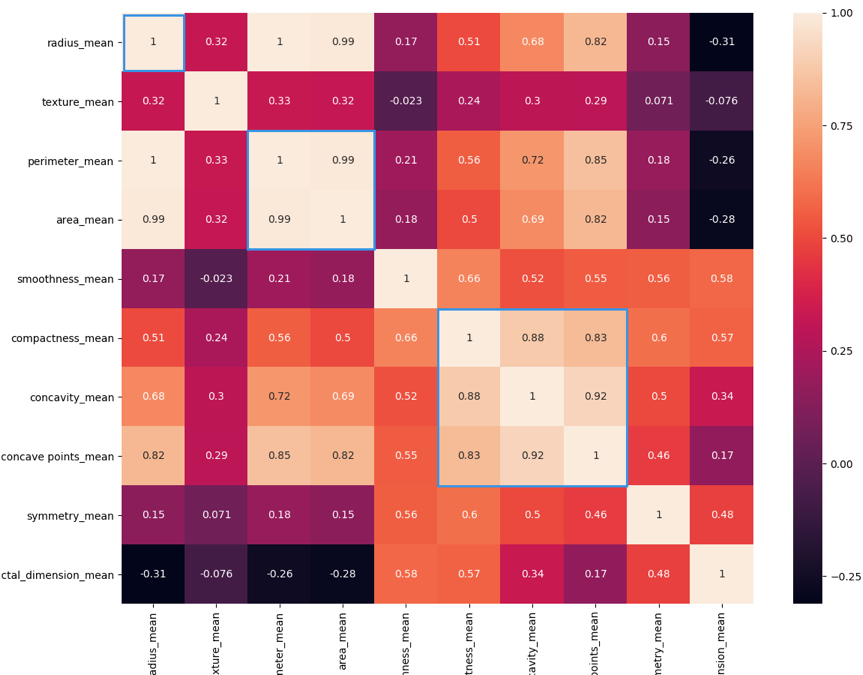
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 import pandas as pdimport matplotlib.pyplot as pltimport seaborn as snsfrom sklearn.model_selection import train_test_splitfrom sklearn import svmfrom sklearn import metricsfrom sklearn.preprocessing import StandardScaler""" 代码来自:https://github.com/cystanford?tab=repositories """ data = pd.read_csv("./breast_cancer_data.csv" ) pd.set_option('display.max_columns' , None ) print(data.columns) print(data.head(5 )) print(data.describe()) features_mean= list(data.columns[2 :12 ]) features_se= list(data.columns[12 :22 ]) features_worst=list(data.columns[22 :32 ]) data.drop("id" ,axis=1 ,inplace=True ) data['diagnosis' ]=data['diagnosis' ].map({'M' :1 ,'B' :0 }) sns.countplot(data['diagnosis' ],label="Count" ) plt.show() corr = data[features_mean].corr() plt.figure(figsize=(14 ,14 )) sns.heatmap(corr, annot=True ) plt.show() features_remain = ['radius_mean' ,'texture_mean' , 'smoothness_mean' ,'compactness_mean' ,'symmetry_mean' , 'fractal_dimension_mean' ] train, test = train_test_split(data, test_size = 0.3 ) train_X = train[features_remain] train_y=train['diagnosis' ] test_X= test[features_remain] test_y =test['diagnosis' ] ss = StandardScaler() train_X = ss.fit_transform(train_X) test_X = ss.transform(test_X) classifier_model = svm.SVC(C = 1.0 , kernel = 'rbf' , degree=3 , gamma='auto' ) classifier_model.fit(train_X, train_y) prediction = classifier_model.predict(test_X) print ("准确率: " , metrics.accuracy_score(prediction, test_y))
这是运行的结果:
img
img
其中的特征选择问题 :
热力图中对角线上的为单变量自身的相关系数是 1。颜色越浅代表相关性越大。所以你能看出来 radius_mean、perimeter_mean 和 area_mean 相关性非常大,compactness_mean、concavity_mean、concave_points_mean 这三个字段也是相关的,因此我们可以取其中的一个作为代表。
特征选择的目的是降维,用少量的特征代表数据的特性,这样也可以增强分类器的泛化能力,避免数据过拟合。
所以我们可以将相关性较强的一组特征视为一类,然后选出一个特征代表该类 。
我们能看到 mean、se 和 worst 这三组特征是对同一组内容的不同度量方式,我们可以保留 mean 这组特征,在特征选择中忽略掉 se 和 worst。同时我们能看到 mean 这组特征中,radius_mean、perimeter_mean、area_mean 这三个属性相关性大,compactness_mean、daconcavity_mean、concave points_mean 这三个属性相关性大。我们分别从这 2 类中选择 1 个属性作为代表,比如 radius_mean 和 compactness_mean。
这样我们就可以把原来的 10 个属性缩减为 6 个属性,代码如下:
1 features_remain = ['radius_mean' ,'texture_mean' , 'smoothness_mean' ,'compactness_mean' ,'symmetry_mean' , 'fractal_dimension_mean' ]